[title]总览[/title]
这篇文章用来解决解卷积的问题。由于基于10X技术的空间转录组(ST)只能做到spot level的分辨率,每一个spot的大小是55um(2021.04.24),每一个spot的基因表达可以理解为是多种细胞类型对应的基因表达的混合,所以如何根据基因表达谱,把一个spot表示成多个细胞类型的混合,就是一个计算问题,这类问题就被称作解卷积(deconvolution)。而目前又有大量缺失了空间结构的单细胞数据,所以把ST和单细胞数据整合在一起,就可以得到每个spot对应的细胞类型。
在上述背景下,这篇文章利用图卷积神经网络来进行解卷积任务,我认为主要有以下几个亮点:
- 利用了图卷积神经网络,考虑单细胞数据到空间转录组之间的非线性关系。
- 成功的把一个无监督或者说是一个描述事物关系的任务转换为了自监督的任务,并且取得了很好的效果。这也是我觉得最有意思的一点。
- 细节的处理很有意思,引入了(典型关联分析)CCA做预处理。
[title]实现思路[/title]
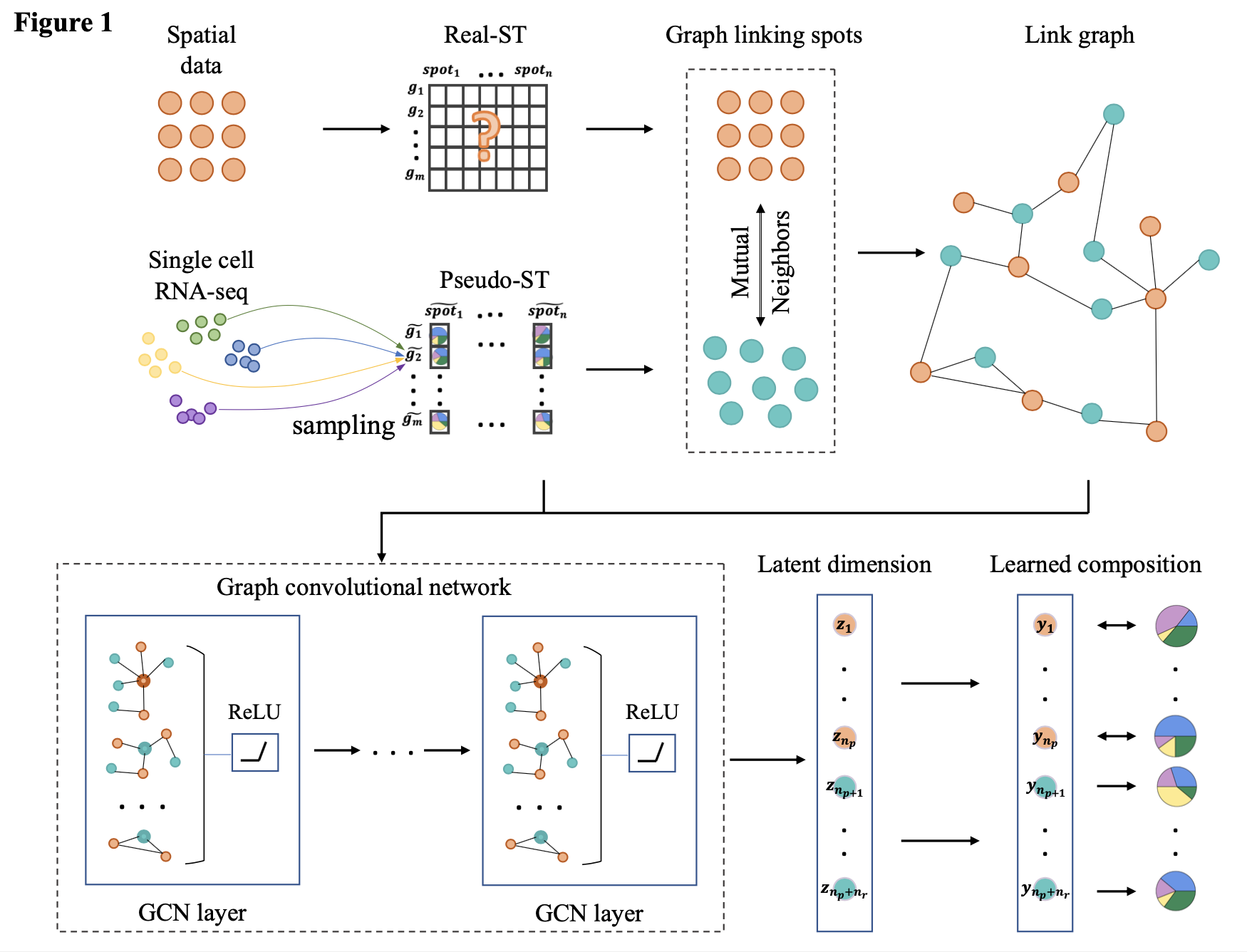
整体的框架如上所示。首先针对单细胞数据,我们知道每个细胞对应的的细胞类型。随机将其中2-8个细胞的基因表达值加在一起,形成一个pesudo spot。考虑到不同细胞的count值不同,采样过程中得到pesudo spot的总count值也不一样,所以DSTG中,每个spot的总count值需要归一化到20000。之后重复上述过程,我们就会得到一个pesudo spot的基因表达矩阵。
然后DSTG利用ANOVA检验找到pesudo spot基因表达矩阵的前2000个HVG。并在ST数据上用同样的方法找到2000个HVG。但注意这时候找到的这两组HVG是不一样的,只是在维度上相同。做完特征筛选,为了继续对两组样本进行降维并且尽可能的保留这两个样本之间的联系,就需要CCA大显身手了。
CCA叫做典型关联分析,简单来讲是一种考虑了两组样本相关性的一种降维方法。比如现在对于样本组X和样本Y,CCA可以在把所有样本都降到1维的同时让样本组X和样本组Y之间的协方差最大(抱歉博客目前还不支持输入公式)。更多细节请参阅典型关联分析(CCA)原理总结 - 刘建平Pinard - 博客园 (cnblogs.com)
总的来说,DSTG先对pesudo 和real spot的表达中每一个基因的表达都进行了归一化,然后利用CCA,将pesudo spot和real spot的特征都降到20维,然后基于KNN,建立所有spot(真的和假的)之间的网络图。为了充分利用真实ST数据的信息,他们还考虑了真实ST数据之间的互近邻关系。最后就得到了一个以所有spot为节点,以0,1表示相似度的网络图了。
具体图神经网络(GCN)的入门不在这篇文章的讨论范围内。上面提到的网络图就是GCN的一个输入,而另一个输入,图上每个节点的特征,就是每个spot的前2000个HVG。这个图神经网络的任务是预测pesudo spots的细胞类型,根据我刚刚提到的,我们是知道Pesudo spot的真实细胞类型组合的,所以这是一个监督学习的问题。对于一个3层的DSTG,网络可以表示为这个式子:
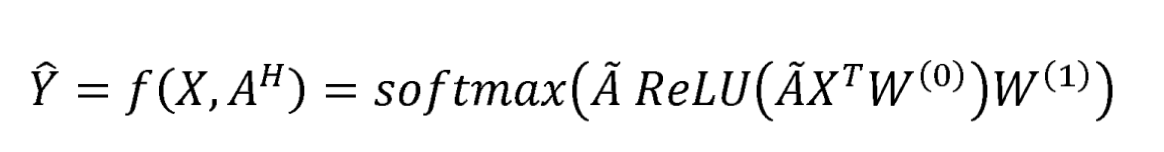
W^0和W^1就是GCN的权重,A是正则化的图(这是图神经网络很常见的trick),最后Y的输出就是一个N*F的向量,N是所有pesudo 和real spots的总和,F是预测的细胞类型分布。最终的Loss函数计算的是pseudo spot预测的细胞类型分布Y和真实分布的交叉熵。
注意在代表GCN的式子中,W^1是h*F的,h是节点在隐层特征的维度,所以可学习的W在所有spot上是一致的。换句话说,当GCN对已知spot的细胞类型预测的很好时,就说明W已经被学好了,就能够预测没有标签的那些spot的细胞类型了。
[title]衡量方法[/title]
为了衡量预测的细胞类型分布与pesudo spot对应真实分布的差异,他们使用了Jensen–Shannon divergence (JSD) score,是KL散度加和与平滑后的形式,这里具体还不太懂,以后有机会研究一下。不过值得注意的是JSD score在另一篇解卷积文章SPOTlight中也被拿来做衡量标准了。
[title]启示[/title]
我觉得是一篇用图卷积用的很巧的方法,化非监督问题为监督问题。但同时,一些细节的处理也很值得学习,比如在对节点降维时没有使用简单的PCA,并且在CCA之前也对特征进行了归一化。
文章评论